《生物信息学分析实践》,作者吴祖建,高芳銮,沈建国,由科学出版社于2010-6-1 出版。描述的是生物信息学简介 。
生物信息学分析_生物信息学分析实践 -图书信息
生物信息学分析实践
作者:吴祖建,高芳銮,沈建国 主编出 版 社: 科学出版社
出版时间: 2010-6-1
开本: 16开
I S B N : 9787030278319
所属分类: 图书 >> 自然科学 >> 生物科学 >> 普通生物学
定价:¥30.0
生物信息学分析_生物信息学分析实践 -目录
序
前言
第一章 生物信息学简介
1.1生物信息学基础
1.1.1 什么是生物信息学
1.1.2 生物信息学的形成与发展
1.1.3 生物信息学的研究内容
1.2 生物信息学应用
1.2.1 生物信息学的热点领域
1.2.2 信息技术与生物信息学
第二章 数据库检索
2.1 综合性数据库NCBI
2.1.1 NCBI简介
2.1.2 NCBI数据库介绍
2.1.3Entrez简介
2.1.4 Entrez检索实例
2.2 综合性数据库EMBL-EBI
2.2.1 EBI简介
2.2.2EBI数据库简介
2.2.3 SRS简介
2.2.4 SRS检索实例
2.2.5 BioMart简介
2.2.6 BioMart检索实例
2.3 UCSC基因组浏览器
2.3.1 UCSC基因组浏览器简介
2.3.2 UCSC基因组浏览器检索实例
第三章 引物设计及测序结果分析
3.1 引物设计
3.1.1 概述
3.1.2 常规PCR引物设计实例分析
3.1.3简并引物设计
3.2 测序结果分析
3.3序列拼接实例分析
第四章 核酸序列分析
4.1 常规分析
4.1.1 核酸序列的检索
4.1.2 核酸序列组分分析
4.1.3 序列变换
4.1.4 限制性酶切分析
4.2 比对分析
4.2.1 BLAST比对
4.2.2 双序列比对
4.2.3多序列比对
4.3 基因结构识别
4.3.1 ORF识别及其可靠性验证
4.3.2 启动子及转录因子结合位点分析
4.3.3 重复序列分析
4.3.4 CpG island
4.3.5 3'UTR区
第五章 蛋白质序列分析
5.1 蛋白质序列的基本性质分析
5.1.1 理化性质分析
5.1.2 疏水性分析
5.1.3 跨膜区分析
5.1.4 信号肽预测
5.1.5 Coil区分析
5.1.6 亚细胞定位“
5.2 结构域分析及motif搜索
5.2.1 结构域分析
5.2.2 motif搜索
5.3 空间结构预测
5.3.1蛋白质二级结构预测
5.3.2蛋白质三级结构预测
5.3.3 蛋白质结构预测方法评价
5.4 抗原表位预测分析
5.4.1 B淋巴细胞抗原表位预测分析
5.4.2 T淋巴细胞抗原表位预测分析
第六章分子进化与系统发育分析
6.1 进化的分子基础
6.1.1 进化树与分子系统学
6.1.2 相似性与同源性
6.1.3 分子进化
6.2 系统发育分析
6.2.1 DNA和氨基酸序列的进化演变
6.2.2系统发育树的种类
6.2.3 用于系统发育分析的分子标记选择
6.2.4 常用的构树方法及其甄选
6.2.5 系统发育分析常用软件
6.3 系统发育分析的检验
6.3.1 系统发育分析方法可靠性的评价
6.3.2 系统树的误差分析及消除方法
6.4 系统树的评估
6.5 系统发育分析实例
6.5.1 使用MEGA软件重建NJ树
6.5.2 使用PAUP软件重建NJ树
6.5.3 使用MEGA软件重建MP树
6.5.4 使用PAUP软件重建MP树
6.5.5 使用PAUP软件重建ML树
6.5.6 贝叶斯树
第七章 RNA分析
7.1 RNA简介
7.1.1 RNA的结构
7.1.2 RNA的功能
7.1.3 RNA研究进展与展望
7.2 RNA二级结构
7.2.1 RNA的二级结构概述
7.2.2 RNA二级结构的预测方法
7.2.3 RNA结构预测实例分析
7.3 高效siRNA的设计
7.3.1 RNAi的作用机制
7.3.2 siRNA的设计原则
7.3.3 影响RNAi的其他因素
7.3.4 siRNA的设计步骤
7.3.5 siRNA的合成
7.3.6 siRNA干涉效果的评判
7.3.7 siRNA相关数据库介绍
7.3.8 siRNA设计实例分析
7.4 microRNA分析
7.4.1 microRNA作用机制概述
7.4.2 miRNA功能与研究方法
7.4.3 miRNA生物信息学分析
7.4.4 miRNA及其靶基因预测的实例分析
主要参考文献及网址
附录
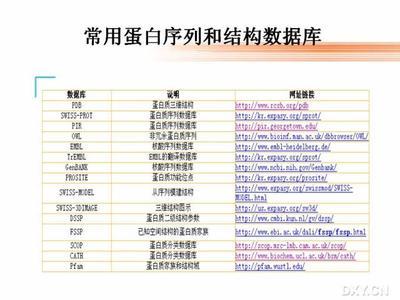
附录1 常用生物学数据库
附录2 各种主要的RNA二级结构预测软件比较
附录3 名词解释
彩图

 爱华网
爱华网