有些网友觉得rna干扰技术论文难写,可能是因为没有思路,所以小编为大家带来了相关的例文,希望能帮到大家!
RNA干扰——理论发展和应用展望篇一
摘要
RNA干扰以及基因沉默是目前分子生物学的一个热门的研究领域,对于基因工程和医药研究的发展有着巨大的潜在推动力。RNA干扰通过对mRNA的特异性抑制从而达到高效的基因沉默,已经显示出了其可能带来医疗技术的突破性发展。本文是对RNA干扰的原理、RNA干扰与基因沉默的理论发展、其在未来可能的应用发展进行的简要综述。
Abstract
RNA interference (RNAi) and Gene Silence are one of the most favorite research fields in molecular biology, RNAi has great potential in the development of the Genetic Engineering and Medical Research. RNAi can make effective Gene Silence through the specific inhibition on mRNA, so it has shown that it can bring a great breakthrough in medical technology. This essay is a brief review on the theory of RNAi and its developments in theories and applications.
正文
I 概述
RNA干扰(RNAi)主要是指外源性的双链RNA(double-stranded RNA, dsRNA)在体内引发的基因沉默(Gene Silence)现象1。RNAi是一种序列特异性的转录后基因沉默机制(post-transcriptional gene silence, PTGS),其在生物体中是普遍存在的2。
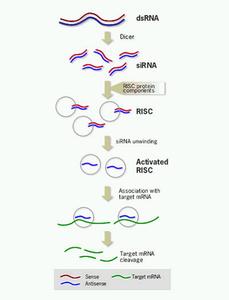
RNAi的作用机制可概括如下:dsRNA进入细胞后,通过ATP依赖性RNA酶III(Dicer, DCR),变为含21-25bp的siRNA(short-interference RNA);siRNA是引发核酸酶降解mRNA的引导物,并与AGO(Argonaute)蛋白结合形成RNA诱导沉默复合体(RNA-induced silencing complex, RISC),RISC对siRNA解链放出反义RNA并与对应的mRNA结合,使其内源性降解;在靶mRNA被降解后,RISC继续靶定其他的mRNA。
同时,有些情况下还会产生扩增效应:siRNA或其反义RNA可通过依赖于RNA的RNA聚合酶(RNA-dependent RNA Polymerase, RdRP)与靶mRNA互补的RNA(cRNA)共同重新合成dsRNA,并通过上述机制生成次级siRNA。通过扩增效应,mRNA的表达会得到完全的抑制,这也是RNAi高效的原因之一。
除了通过siRNA外,在生物体内还发现了另一种RNAi机制:微小RNA(microRNA, miRNA),1
2 RNA干扰:原理与应用,P.3,汤华,科学出版社,2006 RNA干扰技术:从基础科学到药物开发,P.3,(美)Appasani K主编,殷勤伟等译,科学出版社,2007
miRNA是由一种内源性发夹(hairpin)结构转录产物经DCR加工得到的单链RNA,并与AGO蛋白结合生成miRNA-蛋白复合物(miRNA-protein complex, miRNP),通过与靶mRNA结合阻碍其翻译,达到基因沉默的目的。
(图1:RNAi的作用机制)3
RNAi理论虽然是从1990年代才开始发展,但它却逐渐受到人们的重视。1998年在《Nature》上共同发表论文4正式提出RNAi理论的Andrew Fire和Craig C Mello 被授予了2006年诺贝尔生理学或医学奖,以表彰他们在基因技术的使用方面提供了“令人激动的可能性” 、发现了一种有效中止有缺陷的基因运传的机制、这为研发控制这种基因和与疾病作斗争的新药提供了可能性。
从RNAi的作用机制中可以看出,由于siRNA或miRNA对mRNA有着特异性的选择和高效的抑制能力,只要能够确定所要作用的mRNA或对应的基因,就可以针对性地抑制mRNA的基因表达,同时也会将其对其他基因表达的影响降至最低。利用这一点,可以发展很多的治疗技术,用于治疗目前难以治疗的由RNA病毒所引起的疾病,或者一些由于非人体所需的基因表达所导致的疾病比如癌症。
RNAi同样也是一项潜力巨大的基因工程技术。相比较传统的基因敲除、转基因以及突变技术,RNAi更加简易可行,并可用于较大规模的基因功能分析,对于基因工程的实验技术发展有着潜在的巨大推动力。
3 Derek MD, Carl DN, Phillip A. Killing the messenger: Short RNAs that silence gene expression. Nature, 2004, 431: 343~349 4 Fire A, Xu S, Montgomery MK. Potent and specific genetic interference by double-stranded RNA in Caenorhabditis elegans.
Nature, 1998, 391:806~811
II Dicer酶
2001年,Bernstein从果蝇体内分离出一种能够特异性地切割dsRNA的多蛋白核糖核酸酶,并命名为Dicer(DCR)。Bernstein发现一种CG4792酶(Dicer-1)可以将dsRNA全部切割成22个碱基长度的RNA片段,他认为这种酶与RNAi有着很大的相关性5。
DCR的结构中主要包含了三个结构域:以α螺旋为主的RIIIa结构域和RIIIb结构域,以
6及包含α螺旋和β折叠的PAZ结构域。
(图2:鞭毛虫的DCR结构)7
这三个结构域中RIIIa结构域和RIIIb结构域通过切断dsRNA中的磷酸二酯键来将dsRNA剪切为siRNA,而PAZ结构域则在识别dsRNA时起到关键的作用。
在DCR剪切模型的发展中,仍存在着不同的看法。一种是Blaszczyk于2001年提出的包含两个活性中心的DCR模型;另一种是Zhang、Kolb等于2004年提出的包含一个活性中心的DCR模型。
在Blaszczyk模型中,两个活性中心都与Mg2+或Mn2+配位,由二价金属离子辅助剪切dsRNA中的两个磷酸二酯键。该模型是建立在对细菌的Aa-RNase III结构域进行X射线晶体结构研究的基础之上的,发现两个Aa-RNase III可以首尾相连共同剪切一个dsRNA,产生9bp的siRNA产物。8
在Zhang模型中,DCR中只含有一个dsRNA剪切的活性中心,由同一DCR分子中的RIIIa结构域和RIIIb结构域组成。活性中心仍然与Mg2+或Mn2+配位,不过与Blaszczyk模型不同的是,在剪切第二个磷酸二酯键时dsRNA会由原位置旋转约30°,由PAZ结构域重新识别后再剪切。9 5 Bernstein E, Caudy AA, Hammond SM. Role for a bidentate ribonuclease in the initiation step of RNA interference. Nature, 2001, 409: 363~366 6 MacRae IJ, Zhou K, Li F. Structural Basis for Double-Stranded RNA Processing by Dicer. Science, 2006, 311:195~198 7 MacRae IJ, Zhou K, Li F. Structural Basis for Double-Stranded RNA Processing by Dicer. Science, 2006, 311:196-Fig.2 8 Blaszczyk J, Tropea JE. Crystallographic and Modeling Studies of RNase III Suggest a Mechanism for Double-Stranded RNA Cleavage. Structure, 2001, 9:1225~1236 9 Zhang H, Kolb FA. Single Processing Center Models for Human Dicer and Bacterial RNase III. Cell, 2004,
III RISC
RNA诱导沉默复合体(RNA-induced silencing complex, RISC)是在RNAi的最后阶段中起到剪切降解靶mRNA作用的关键反应物。RISC是由siRNA或miRNA与AGO(Argonaute)蛋白形成的复合体,其组装经由RISC装载复合体(RISC loading complex, RLC),使siRNA与AGO蛋白组合。而RISC对靶mRNA的剪切则是通过AGO蛋白的PAZ结构域的识别以及PIWI结构域及剪切完成的。
目前也有研究表明在细胞核中也有RNAi的相关成分参与了DNA甲基化等反应,其中有与RISC类似的RNA诱导的转录性基因沉默启动复合体(RNA-induced transcriptional silencing complex, RITS)的存在11,说明RISC在RNAi中具有重要的地位。
AGO蛋白中的主要活性区域是PAZ和PIWI。PAZ结构域对于3′端单链结构的单链RNA和dsRNA具有强烈的亲和作用,因此可以识别siRNA以及mRNA。而在PIWI结构域中具有一个剪切mRNA的活性中心,可以切断mRNA的磷酸二酯键,在这个活性中心处,siRNA与10 MacRae IJ, Zhou K, Li F. Structural Basis for Double-Stranded RNA Processing by Dicer. Science, 2006, 311:197-Fig.4 11 Craig C. Mello, Darryl Conte Jr. Revealing the world of RNA interference.
mRNA互补配对。
RISC的组装模型至今没有定论,不过基本上都是通过RLC中间复合体进行的,RLC是由剪切dsRNA的DCR和siRNA/miRNA与AGO蛋白结合的中间体,再解离出DCR和RISC。在不同的生物体中,组装的模式和参与组装的底物也不同。 12 Elkayam E, Greene EM. The Structure of Human Argonaute-2 in Complex with miR-20a.
2004年,Song在研究激烈火球菌(Pyrococcus furiosus)AGO蛋白晶体结构的基础上提出了一种可能的RISC剪切模型。他发现在AGO蛋白中PAZ和PIWI结构域中有可能容纳底物的沟槽状结构,PAZ和PIWI分别位于这个沟的两侧,Song推测PAZ识别siRNA后会诱导siRNA以及与其互补的靶mRNA进入沟,并在PIWI的活性中心处将靶mRNA的磷酸二酯键切断,达到抑制靶mRNA表达的作用。
1414 Tang G. siRNA and miRNA: an insight into RISCs. TRENDS in Biochemical Science, 2005, 30:106~114 Song JJ, Smith SK, Hannon GJ. Crystal Structure of Argonaute and Its Implications for RISC Slicer Activity.
IV RdRP
依赖于RNA的RNA聚合酶(RNA-dependent RNA Polymerase, RdRP)普遍存在于真菌、植物以及线虫中,对于这些生物体中的RNAi是必须的,RdRP可以以siRNA为底物合成新的dsRNA。
一种典型的RdRP的结构图,其蛋白质结构中主要是α螺旋结构。RdRP的作用是将游离的siRNA与靶mRNA结合,并以此为模板聚合成新的dsRNA。而新的dsRNA再通过DCR剪切形成siRNA(次级siRNA),次级siRNA又可以通过RdRP再一次进行该反应,因此可以通过这种扩增机制产生大量的siRNA,使得靶mRNA得到完全抑制。
这里值得注意的一点是,由于RdRP用siRNA和靶mRNA为模板,而siRNA的长度明显小于靶mRNA,因此合成的dsRNA被DCR剪切形成的次级siRNA不仅仅是原siRNA,还会包含原siRNA的上游序列或者下游序列。这些次级siRNA所靶定的mRNA的区域可能并非原来所期望的区域,因此产生了过渡性RNAi(transitive RNAi)。不过,所有的次级siRNA所抑制降解的仍是原靶mRNA,因此过渡性RNAi并不会使RNAi发生错误。
V RNAi在医疗中的应用
由于RNAi在疾病预防和治疗领域表现出巨大潜力,从这一现象发现开始,就不断有大量的研究将RNAi应用于医疗,虽然RNAi仍然是一个比较新的领域,但是其在医疗方面的应用已经出现了不少令人期待的成果。
目前RNAi已被应用于HIV病毒的预防的治疗,在2008年,Priti Kumar在《Cell》上发表论文称观察到细胞外导入的siRNA可以有效地抑制HIV-1病毒对T细胞的感染17。
2002年,Lassus等人用RNAi技术分别沉默了Caspase-1和Caspase-2的基因表达,发现Caspase-2蛋白质水平降低后可以降低由于药物造成DNA损伤导致的细胞凋亡18。由于细胞凋亡与器官功能衰退以及恶性肿瘤的形成有着密切的联系,因此RNAi技术也有望应用于治疗器官衰竭和恶性肿瘤。
VI RNAi在基因工程中的应用
目前已经发现了4种在细胞基因组水平发挥作用的dsRNA介导途径,分别为RNA指导的DNA甲基化(RNA-directed DNA methylation, RdDM)、RNAi介导的异染色质的形成、DNA切除以及减数分裂性沉默19。
应用RNAi可以将特定的mRNA的基因表达沉默,因此很适合用于遗传学研究,只要筛选出靶mRNA对应的siRNA,就可以通过细胞外导入siRNA来达到基因沉默的目的,这样可以很方便地观察某个基因的功能,同时也不会对其他的基因和细胞功能产生影响。
16 Nishikura K. A Short Primer on RNAi: RNA-Directed RNA Polymerase Acts as a Key Catalyst. Cell, 2001,107:415~418 17 Kumar P, Kim SS. T Cell-Specific siRNA Delivery Suppresses HIV-1 Infection in Humanized Mice. Cell, 2008, 134:577~586 18 Lassus P. Requirement for Caspase-2 in Stress-Induced Apoptosis Before Mitochondrial Permeabilization. Science, 2002, 297:1352~1354 19 RNA干扰:原理与应用,P.124,汤华,科学出版社,
References
I 参考书目
1 RNA干扰:原理与应用,汤华,科学出版社,2006
2 RNA干扰技术:从基础科学到药物开发,P.3,(美)Appasani K主编,殷勤伟等译,科
学出版社,2007
II 参考文献
1 Derek MD, Carl DN, Phillip A. Killing the messenger: Short RNAs that silence gene expression. Nature, 2004, 431: 343~349
2 Fire A, Xu S, Montgomery MK. Potent and specific genetic interference by double-stranded RNA in Caenorhabditis elegans. Nature, 1998, 391:806~811
3 Bernstein E, Caudy AA, Hammond SM. Role for a bidentate ribonuclease in the initiation step of RNA interference. Nature, 2001, 409: 363~366
4 MacRae IJ, Zhou K, Li F. Structural Basis for Double-Stranded RNA Processing by Dicer. Science, 2006, 311:195~198
5 Blaszczyk J, Tropea JE. Crystallographic and Modeling Studies of RNase III Suggest a Mechanism for Double-Stranded RNA Cleavage. Structure, 2001, 9:1225~1236
6 Zhang H, Kolb FA. Single Processing Center Models for Human Dicer and Bacterial RNase
III. Cell, 2004, 118:57~68 20 Craig C. Mello, Darryl Conte Jr. Revealing the world of RNA interference.
7 Craig C. Mello, Darryl Conte Jr. Revealing the world of RNA interference. Nature, 2004, 431:338~342
8 Elkayam E, Greene EM. The Structure of Human Argonaute-2 in Complex with miR-20a. Cell, 150:100~110
9 Tang G. siRNA and miRNA: an insight into RISCs. TRENDS in Biochemical Science, 2005, 30:106~114
10 Song JJ, Smith SK, Hannon GJ. Crystal Structure of Argonaute and Its Implications for RISC Slicer Activity. Science, 2004, 305:1434~1437
11 Hansen JL, Long AM, Schultz SC. Structure of the RNA-dependent RNA polymerase of poliovirus. Structure, 1997, 5:1109~1122
12 Nishikura K. A Short Primer on RNAi: RNA-Directed RNA Polymerase Acts as a Key Catalyst.
Cell, 2001,107:415~418
13 Kumar P, Kim SS. T Cell-Specific siRNA Delivery Suppresses HIV-1 Infection in Humanized Mice. Cell, 2008, 134:577~586
14 Lassus P. Requirement for Caspase-2 in Stress-Induced Apoptosis Before Mitochondrial Permeabilization. Science, 2002, 297:1352~1354
15 Meister G, Tuschl T. Mechanisms of gene silencing by double-stranded RNA. Nature, 2004 431:343~349
16 Bartel DP. MicroRNAs: Genomics, Biogenesis, Mechanism, and Function. Cell, 2004, 116:281~297
17 Tijsterman M, Ronald H.A. Plasterk. Dicers at RISC: The Mechanism of RNAi.
RNA干扰技术论文篇二
RNA干扰(RNA interference, RNAi)是指在进化过程中高度保守的、由双链RNA(double-stranded RNA,dsRNA)诱发的、同源mRNA高效特异性降解的现象。它是生物进化过程中遗留下来的一种在转录后通过RNA调控基因表达的机制。最早的RNA干扰研究是从植物和线虫开始,2001年Tuchl等首次应用长度约为19~23个碱基对的双链小分子干扰RNA(small interfering RNA,siRNA)在哺乳动物细胞中诱发基因沉默现象,证实这些细胞也普遍存在RNA干扰的机制,从而在世界范围内掀起了研究和应用RNA干扰技术的热潮。
RNA干扰之所以能引起生物医学界几乎所有研究领域的广泛性趣,是因为siRNA具有强大的抑制基因表达的效应和高度的序列特异性。与抑制基因表达的传统工具,如反义寡核苷酸和核酶等比较,siRNA沉默基因的效率高达数十到数千倍,是逆基因工具的革命性改进。此外与目标基因信使RNA相差一个碱基序列的siRNA的基因沉默效应大大受到削弱,从而保证了抑制目标基因的高度特异性。因此,siRNA的发现具有划时代的意义,它不仅深入揭示了细胞内基因沉默的机制,而且它还是基因功能分析的有力工具,极大地促进了人类揭示生命奥秘密的进程。siRNA本身还是一种极具前景的基因靶向药物,可广泛地用于诸如癌症等疾病的治疗。鉴于siRNA技术的巨大意义与广阔应用前景,siRNA技术连续多年被美国《Science》杂志评选的“全球年度十大科学突破”。
由于使用RNAi技术可以特异性剔除或关闭特定基因的表达,所以该技术已被广泛用于探索基因功能和传染性疾病及恶性肿瘤的基因治疗等领域。
第一节 RNA干扰的研究历程
虽然RNA干扰是一种古老的机制,但是第一篇报道这种现象的论文是在1990年发表的。
1990年,Napoli和Van der Krol等在研究矮牵牛花查尔酮基因时发现了基因共抑制现象(cosuppression)。她在体外构建了控制矮牵牛花花色的基因查尔酮基因片段,连上花椰菜花叶病毒的35S启动子,连入农杆菌的T-DNA质粒,在矮牵牛花中过度表达。她原先预期,查尔酮的过度表达能加深牵牛花花色的紫色,但是结果却导致了矮牵牛花子代花色的褪色,内源性的查尔酮遭到沉默。Napoli教授把这一现象称之为内源性基因共抑制现象。 1998年,Andrew J. Hamilton教授研究了番茄ACC氧化酶基因的基因共抑制现象。他利用放射性标记法和放线菌酮处理法分析转录后ACC氧化酶基因的mRNA和成熟mRNA。实验结果表明,外源重组基因片段能成功转录,但是mRNA成功转录后,因为某种原因发生了转录后基因沉默(post transcriptional gene silencing,PTGS)。因此,Andrew J. Hamilton认为内源性基因共抑制现象属于转录后的基因沉默现象。
1995年,Guo等发现注射正义RNA(sense RNA)和反义RNA(antisense RNA)均能有效并特异性地抑制秀丽新小杆线虫(C. elegans)par-1基因的表达,该结果不能使用反义RNA技术的理论做出合理解释。直到1998年,Fire和Mello课题组接手了此课题。他们以秀丽新小杆线虫为模型,发现在Guo的课题中,引发线虫par-1基因沉默的是小片段的双链RNA,而不是正义单链RNA或负义单链RNA。他们之后又研究了秀丽新小杆线虫的unc-22基因,进一步阐述了双链RNA在基因沉默中的作用,并将这一现象命名为“RNA干扰”。 他们的研究成果激起了其他科学家研究RNA干扰现象的浓厚兴趣,由于他们的发现揭示了分子生物学中一个全新的,具有普遍性的机制,Andrew Fire, Craig C. Mello两位科学家因此在2006年获得诺贝尔奖。
此后dsRNA介导的RNAi现象陆续发现于真菌、果蝇、拟南芥、锥虫、水螅、涡虫、斑马鱼等多种真核生物中,并逐渐证实植物中的转录后基因沉默、共抑制(cosuppression)及RNA介导的病毒抗性、真菌的抑制(quelling)现象均属于RNA干扰在不同物种的表现形式。
第二节 RNA干扰原理
病毒基因、人工转入基因、转座子等外源性基因随机整合到宿主细胞基因组内,并利用宿主细胞进行转录时,常产生一些dsRNA(图1)。宿主细胞对这些dsRNA迅即产生反应,其胞质中的核酸内切酶Dicer将dsRNA切割成多个具有特定长度和结构的小片段RNA(大约21~23 bp),即siRNA。研究表明在生物体中siRNA具相似的结构特征:为长约21~23bp的双链RNA,具5’单磷酸和3’羟基末端,互补双链的3’端均有一个2~3nt的单链突出。负责将dsRNA转化为siRNA的Dicer核酸酶,属于RNaseⅢ家族,具有两个催化结构域、一个解旋酶(helicase)结构域和一个PAZ (Piwi/Argonaute/Zwille)结构域,Dicer在催化过程中以二聚体的形式出现,其催化结构域在dsRNA上反平行排列,形成四个活性位点,但只有两侧的两个位点有内切核酸酶活性,这两个位点在相距约22bp的距离切断dsRNA,各种生物体内Dicer结构略有不同,致使siRNA长度存在微小差别。
siRNA在细胞内RNA解旋酶的作用下解链成正义链和反义链,继之由反义siRNA再与体内一些酶(包括内切酶、外切酶、解旋酶等)结合形成RNA诱导的沉默复合物(RNA-induced silencing complex,RISC)。RISC与外源性基因表达的mRNA的同源区进行特异性结合,RISC具有核酸酶的功能,在结合部位切割mRNA,导致特定基因沉默,切割位点即是与siRNA中反义链互补结合的两端。被切割后的断裂mRNA随即降解,从而诱发宿主细胞针对这些mRNA的降解反应。
在RISC中,起靶序列识别作用的是siRNA的反义链,Zamore等发现在RNAi过程中,首先产生的是RISC无活性前体,分子量250kD,当加入ATP后可形成100kD的活性复合体。由无活性前体向活性酶复合物的转换类似蛋白酶原的激活,但RISC酶复合物激活不需要共价键断裂,而要求结合于其上的siRNA双链的解开。在ATP存在时,依赖于ATP的解旋酶解开siRNA的双链并将其正义链与靶mRNA置换,mRNA取代正义链与反义链互补,然后由活化的RISC在互补区的中间,距离siRNA反义链3’末端约12bp处切断靶mRNA序列。
siRNA不仅能引导RISC切割同源单链mRNA,而且可作为引物与靶RNA结合并在RNA聚合酶(RNA-dependent RNA polymerase,RdRP)作用下合成更多新的dsRNA,新合成的dsRNA再由Dicer切割产生大量的次级siRNA,经过若干次的合成-切割循环,RNAi的作用不断放大,最终将靶mRNA完全降解。
此外许多研究还显示RNAi信号可以越过胞间屏障向其他细胞和组织扩散,在植物中,RNAi信号可以通过两种途径在细胞间传递:①短距离的相邻细胞之间的传递,植物细胞之间有着非常丰富的连接——胞间连丝,沉默信号(如siRNA分子)可以通过胞间连丝在细胞间传递;②沉默信号还可以通过植物里纵横交错的脉管系统进行长距离的传送。而在动物中,RNAi信号的扩散需要特殊的蛋白参与,Hunter等在线虫中鉴定出了一种与沉默信号传播相关的蛋白,它是由SID一1基因编码的一种跨膜蛋白,能在膜上形成跨膜通道供沉默信号通过,SID-1蛋白同源物在果蝇中不存在,但有研究表明在哺乳动物中存在SID-1蛋白同源物。
RNAi发生于除原核生物以外的所有真核生物细胞内。需要说明的是,由于dsRNA抑制基因表达具有潜在高效性,任何导致正常机体dsRNA形成的情况都会引起不需要的相应
基因沉默。所以正常机体内各种基因有效表达有一套严密防止dsRNA形成的机制。
怀特黑德生物医学研究所的本杰明·刘易斯等研究人员发现小RNA能通过阻断蛋白质合成的方式调控基因表达。他们借助一个计算模型来确定小RNA和对应的基因,发现了miRNA控制很大一部分生命功能的证据。
研究人员比较了人类和狗、鸡、鼠的基因组,对这几个物种共有的蛋白质合成基因与miRNA寻求对应关系。结果发现,尽管这几个物种在3.1亿年前就开始“分家”各自进化,但它们基因组中受miRNA调控的基因都占三分之一左右,而且这些基因在进化过程中都得以保存而未发生变化。刘易斯说,随着更多的基因组数据发布以及实验技术的进步,还可能发现更多的基因是由小RNA调控的。
线虫中的RNA干扰模型
Tabara教授于1999年以秀丽新小杆线虫为模型(图2),揭示了RNA干扰的原理。线虫细胞上的跨膜蛋白SID-1引导细胞外的长链双链RNA进入细胞质,RNA立即与细胞质内的RDE-4,RDE-1蛋白形成复合体。然后DCR-1与DRH-1蛋白与该复合体结合,DCR-1蛋白将长链RNA切断,得到RDE-1结合的siRNA复合物。RDE-1-si-RNA复合物在解旋酶的作用下解链,RDE-1蛋白脱去,RISC酶与正义链和反义链中的一条链结合,RISC-单链RNA复合物在引导下与靶mRNA结合。反义RNA与靶mRNA上同源的部分特异性结合,在RISC的作用下,靶mRNA链被切断,无法与核糖体结合,翻译成蛋白质,引起基因沉默。同时,以反义RNA为引物,以mRNA为模板,在RdRP的作用下,生成新的siRNA反义链。这就是siRNA的级联放大的机制。
RNA干扰现象介导的mRNA降解特异性强,同时由于放大机制的存在微量siRNA就能引起明显的RNA干扰现象。线虫中的RNA干扰机制比较典型,但是在高等生物中,RNA的机制又有细微差别。
随着RNAi技术的不断成熟,尤其在哺乳动物细胞中应用和载体转染siRNA成功后,对肿瘤的研究有了前所未有的突破,研究的重点主要在以下几个方面:
(1)癌基因及抑癌基因的敲除:肿瘤是机体遗传和环境致癌因素以协同或序贯的方式,引起遗传物质DNA损伤、突变,同时伴随有多个癌基因激活和抑癌基因失活,使正常细胞不断增生、转化而形成的新生物。因此,如果可以抑制体内原癌基因的激活或者增强抑癌基因的活性,那么肿瘤的发生率就会降低,甚至可以永久地避免相关肿瘤的发生。
Brummelkamp等用病毒载体将siRNA导入人的胰腺癌细胞,可特异且稳定地抑制癌基因K-RASV12的表达,这个癌基因的受抑使肿瘤的生长能力及恶性程度降低Yoshinouchi等向体外培养的SiHa宫颈癌细胞内导入癌基因E6的siRNA后,发现SiHa细胞的生长受到抑制,且随着E6癌基因的受抑,E7癌基因的量也下降;而p53蛋白的量增高,Rb蛋白的磷酸化程度也随之降低,表明抑癌基因也受到了激发。更令人兴奋的是,导入E6基因siRNA的SiHa细胞体内接种后,较未受抑制的对照组生长明显缓慢,甚至停止生长,这给在体基因治疗奠定了坚实的基础。Shen等首次应用重组了siRNA的腺病毒载体,在人类乳腺癌细胞和人类肺癌细胞A549中成功抑制了抑癌基因p53和MCF-7。人类9号和22号染色体的置换使22号染色体上的BCR基因与9号染色体上的ABL基因融合,产生的BCR-ABL融合蛋白诱发酪氨酸激酶强激活而致白血病发生。Druker等在设计干扰基因的位点时选取了融合位点,使白血病细胞出现凋亡。
对基因的某个部分用siRNA进行特异性敲除也成为研究基因功能的一个重要手段,敲除后,可通过观察细胞或生物基因性状的改变来研究该段基因在生物体内的真正作用。如神经元泛肽C末端水解酶L1(neuronal ubiquitin C-terminal hydrolase,UCH-L1)在肺癌的某种细胞内表达,而在正常的肺组织中却没有该物质。Liu等在研究UCH-L1的作用时选择了用RNAi来抑制,发现抑制了UCH-L1后肿瘤细胞反而生长旺盛,表明UCH-L1可能抑制肿瘤细胞的生长。在研究前列腺癌的过程中,Varambally等发现了一种EZH2蛋白,其在已发生转移的前列腺癌细胞中表达的量比正常组织细胞中高得多,因此可以作为一种肿瘤的标记物;而进一步用siRNA抑制该基因,可以使前列腺癌细胞的生长明显受抑,提示EZH2可以作为肿瘤的靶点试用于前列腺癌的临床治疗。
bcl-2基因的主要作用是抑制细胞凋亡和延长细胞的存活。Lin等在人的前列腺癌细胞LNCaP中,用cDNA和mRNA寡核苷酸混合物D-RNAi抑制blc-2基因导致了基因静默,并使细胞凋亡。他们认为,前列腺癌细胞的blc-2高表达可以使肿瘤的抗凋亡能力增强,这为RNAi可能治疗前列腺癌提供了依据。对食管腺癌的研究发现,blc-xL基因也与细胞的抗凋亡有关,食管腺癌细胞表达blc-xL基因的能力明显增强。体外培养的细胞转染siRNA后,blc-xL在mRNA水平减少了60%,在蛋白质水平减少了50%。因此,细胞的抗凋亡能力也随之下降。
(2)有关肿瘤性疾病信号传导途径的研究:Crans Vargas等用siRNA抑制了环磷酸腺苷反应元件结合蛋白(cyclic adenosine monophosphate responseelement binding protein,CREB),这种蛋白可能在急性白血病的信号传导过程中起作用,抑制了该基因后,培养的白血病细胞生存数量明显减少。我们还可以利用siRNA抑制一些肿瘤信号传导途径中的必要因素,来达到治疗肿瘤的目的。
(3)肿瘤免疫逃逸方面的研究:人体自身的细胞毒T淋巴细胞(cytotoxic T lymphocyte,CLT)要杀伤肿瘤细胞必须靠靶细胞的主要组织相容性复合体-Ⅰ(major histocompatibility complex,MHC-Ⅰ)类分子的识别,如果MHC-Ⅰ类分子在肿瘤的表面表达下降,自身的CTL就无法识别,从而导致肿瘤的免疫逃逸。前髓细胞白血病(promyelocytic leukemia,PML)基因是肿瘤抑制基因,如果PML可以抑制MHC-Ⅰ类分子的表达,那么这种白血病分子就可以逃逸体内的免疫系统,而Bruno等的研究发现,PML与MHC-Ⅰ类分子的表达没有明显关系,用RNAi等方法抑制PML后,预计的MHC-Ⅰ类分子下调并没有出现,提示在人的PML细胞中并未通过PML下调MHC-Ⅰ类分子来逃逸宿主免疫系统。
实体瘤组织由瘤细胞及间质构成,而后者主要以新生血管成分为主,在肿瘤的生长和扩散中起重要作用。血管内皮生长因子(vascular endothelial growth factor,VEGF)家族是已知特异性作用于内皮细胞的生长因子,在血管生成中作用至关重要。Zhang等 在鼠的上皮卵巢癌细胞中,应用载体介导VEGF的siRNA,有效降低了实验鼠的VEGF提示可以利用VEGF在肿瘤的血管生长中的重要作用来治疗肿瘤。RNAi可以特异性抑制肿瘤内的VEGF,而其他药物的特异性远不能及。Bertrand等在体外培养的Hela细胞和异种移植的小鼠体内进行了RNAi与反义核酸抑制的对比实验,结果显示,RNAi的抑制效率远高于反义核酸,而且持续的时间延长;在小鼠体内反义核酸根本就没有作用发生,而RNAi的活性仍然很高,推测反义核酸可能被体内的核酸降解系统破坏了。Niu等在血管生成信号传递的过程中,用RNAi抑制Tie-2后,血管内皮细胞的生存能力下降,而且还导致了Akt信号系统受阻,结果使血小板反应蛋白的量增加。血小板反应蛋白是一种内源性抗血管生成物质,可以调控肿瘤血管的生成及肿瘤的生长,甚至可以导致内皮细胞的凋亡。
(4)提高肿瘤对化疗及放疗的敏感性:肿瘤化疗失败的主要原因是肿瘤细胞获得了耐药性,这种耐药性常表现为肿瘤细胞对多种结构不同、作用靶位不同、作用方式不同的抗肿瘤药物具有抵抗性,称为多药耐药(multi-drug resistance,MDR)。研究发现,这种耐药性与肿瘤细胞的某些耐药基因相关,主要的耐药基因是多药耐药基因1(MDR1),编码相对分子质量为170 000的跨膜糖蛋白P-gp/P-170。这种糖蛋白具有药泵的作用,当其表达增高或功能异常增强时,能将肿瘤细胞内的药物“泵”出细胞外,使细胞能在高浓度的药物环境中生存。针对该基因的RNAi可以使MDR1基因的水平下调而进一步提高细胞内的药物浓度,提高肿瘤细胞对化学治疗的敏感性。Nieth等在人胃癌细胞EPG85-257RDB和胰腺癌细胞EPP85-188RDP中,应用siRNA成功抑制了MDR1基因及其表达产物,抑制效率达91%,两种细胞对柔红霉素的耐药性抵抗分别降低了58%和89%。表明此种siRNA 也可以试用于肿瘤的治疗,通过提高细胞对抗肿瘤药物的敏感性来达到治疗肿瘤的目的。对于p53基因缺陷的肿瘤细胞,因其G1期的检查位点缺失,所以可以逃逸体内的免疫。Wang等找到了可以在G2期激活检测位点的Chk1、Weel和Myt基因,用siRNA分别抑制这几个基因以后,宫颈癌细胞Hela出现了凋亡,而正常的乳腺细胞却没有出现凋亡。表明Chk1、Weel和Myt基因可以试用于放疗及化疗,以提高对p53基因缺失的肿瘤细胞的疗效。细胞的DNA修复系统能对放射线所致的DNA, 损伤予以修复,从而导致肿瘤细胞对放射治疗的抵抗,而双链DNA打开后的修复错误可以使肿瘤细胞对放射治疗的敏感性提高。DNA活化的蛋白激酶接触反应多肽(PRKDC)参与了DNA双链的修复,如果PRKDC被siRNA抑制,细胞的放射敏感性就能大大提高,就可以通过减少放射剂量来降低放疗的副作用。
目前,RNAi的体内研究逐步深入。Hasuwaa等在转基因的小鼠及大鼠胚胎细胞中注入带有siRNA的载体,发现胚胎中的各个器官均广泛地出现了特异性的抑制现象。Sorensen等通过静脉注射或腹腔注射两种不同的方式分别将带有RNAi的阳离子脂质体植入成年小鼠体内,达到了抑制内源性及外源性基因的目的,并且认为合成的RNAi可以在体内达到与普通药物相同的功能。如果RNAi技术将来能应用于临床,将使肿瘤的治疗变得简单方便。
 爱华网
爱华网